很多朋友表示不太清楚怎么使用DNAMAN对比核酸序列?下面小编就将和大家一起分享关于使用DNAMAN对比核酸序列的方法,有需要的朋友可以来看看哦,希望可以帮助到各位朋友。
方法1:
1.在电脑上打开DNAMAN软件,打开File-New,将序列粘贴到弹出的窗口中,点击File-save,保存到指定的文件夹。
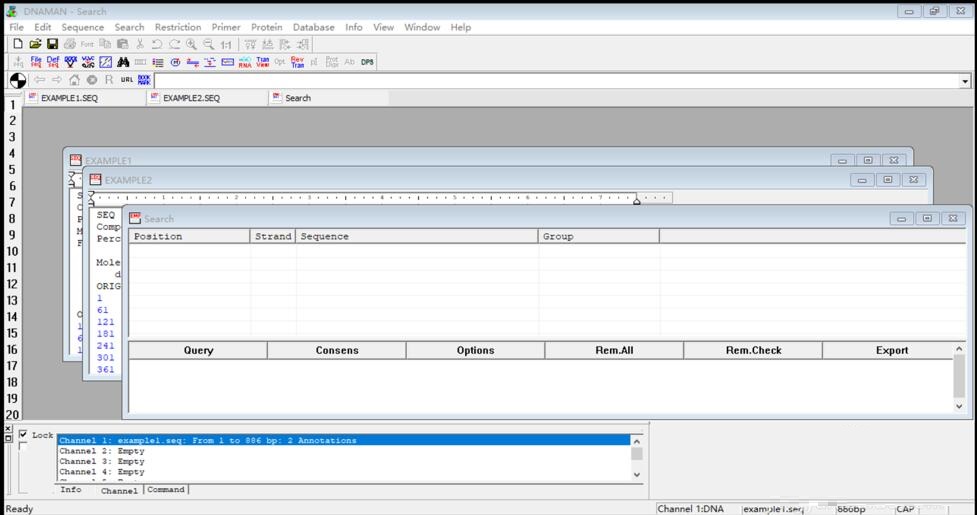
方法2:
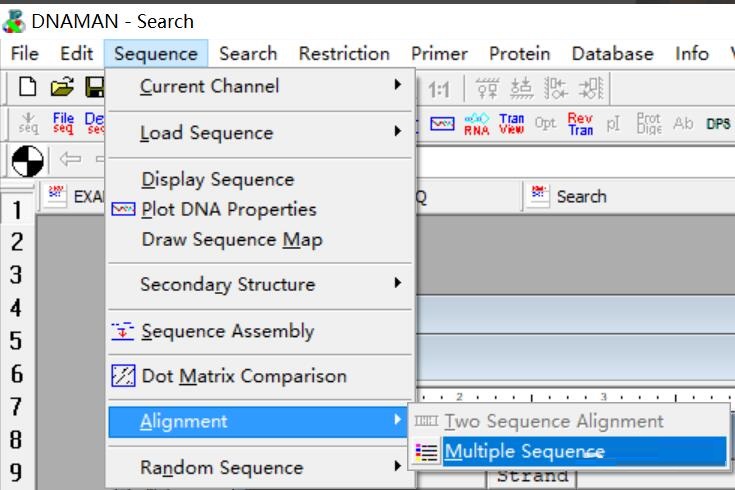
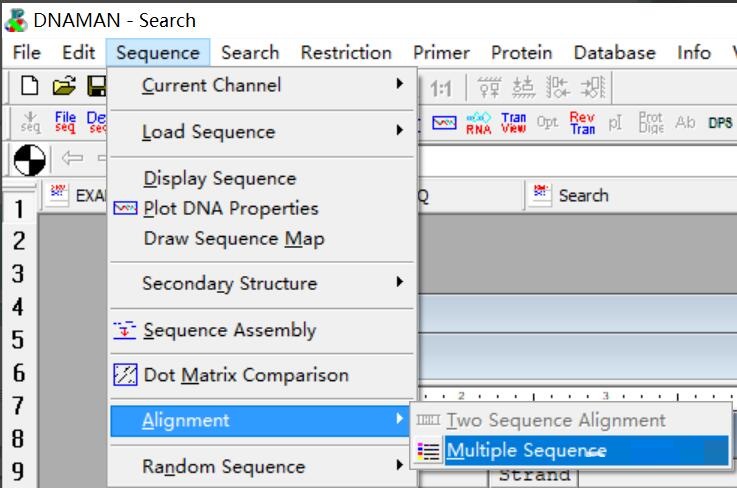
1.将所需比对的序列保存好以后,选中Sequence—Aligment—Multiple aligment sequence进行多序列比较。
2.在弹出的窗口Sequence&Files中加载序列,File、Fold、channel、Database分别表示从文件、文件夹、channel和数据库中获取序列。勾选窗口中的“DNA”,点击“下一步”。
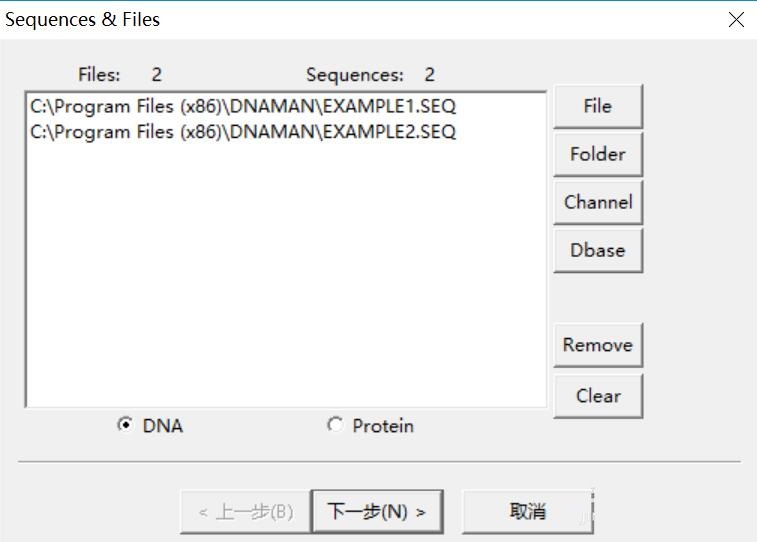
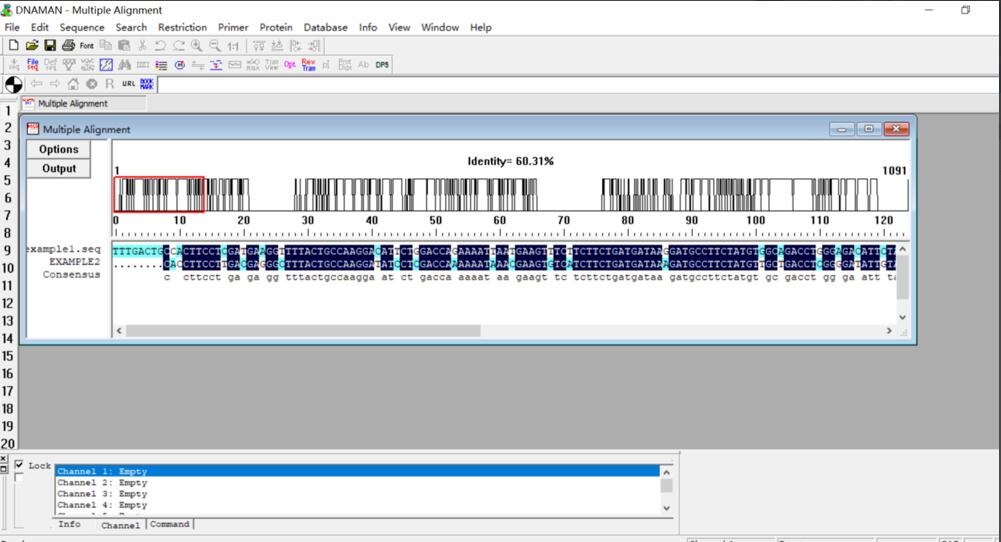
3.在弹出的窗口Method中,“optimalaligment”最佳比对方式中有四个高大上的选项:Full Alignment(完全比对)、 Prosile Aligment(轮廓比对)、 New Swquence on Profile (轮廓上的新序列)、Fast Alignment(快速比对),本文选择了Fast Alignment,并且勾选了Try both strands(尝试使用双链)。其他项目不需修改,默认就OK,点击“下一步”。
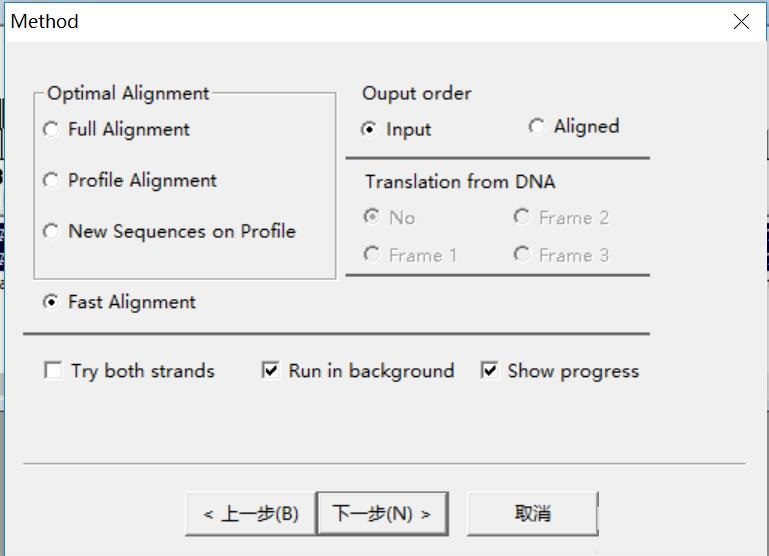
4.“Duang”,结果就出来啦!点击“Option”可以选择要显示的内容。
方法3:
1.保存:点击File-output-Graphic(EMF)File,该序列比对的结果窗口可以图片格式保存。
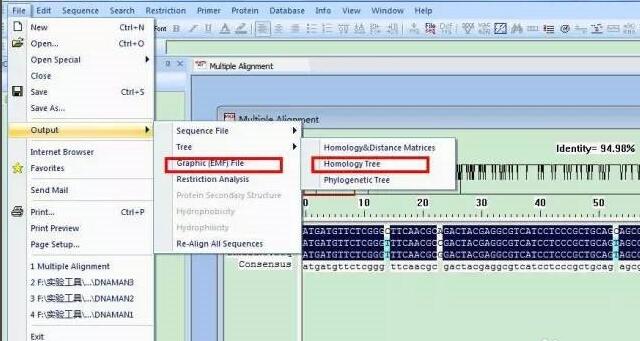
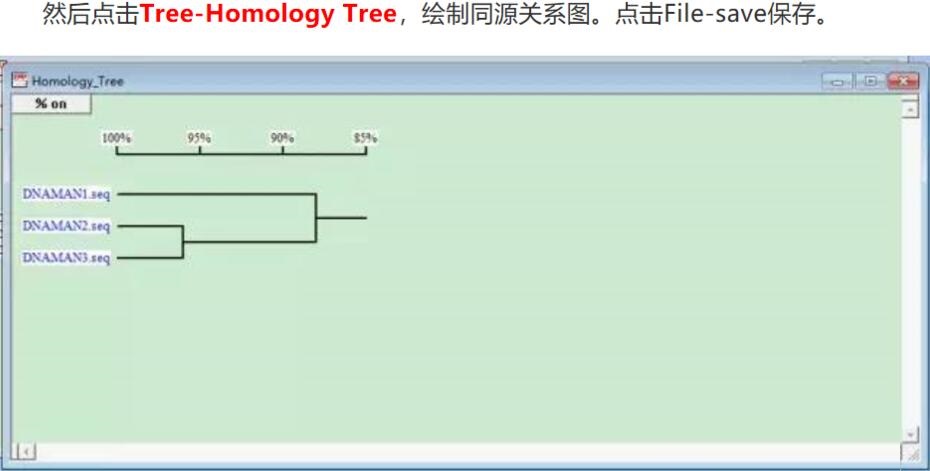
2.做进化树分析,并保存,就完成了。
以上就是小编分享的使用DNAMAN对比核酸序列的方法,还不太清楚的朋友可以来学习一下哦。




